1細胞レベルにおける多細胞組織の動態
多細胞生物の胚組織は三次元的な多細胞構造を有している.この三次元的な組織構造は,発生・代謝・疾患の過程において動的に変化する.特に,胚組織の発生過程では,上皮組織(epithelium)の三次元的な形態形成がよく研究されている.
このような胚組織の構造は,胚・器官・細胞・分子など,大きくスケールの異なる階層性を有している.特に,分子スケールでは,遺伝子発現・シグナル伝達などの生化学的な振る舞いにより,細胞の分化状態や細胞間の相互作用が制御され,組織上における細胞の領域化が行われる.さらに,集団的な細胞の力発生により,細胞の変形・運動が生じ,組織・器官スケールの三次元的な変形が実現される.
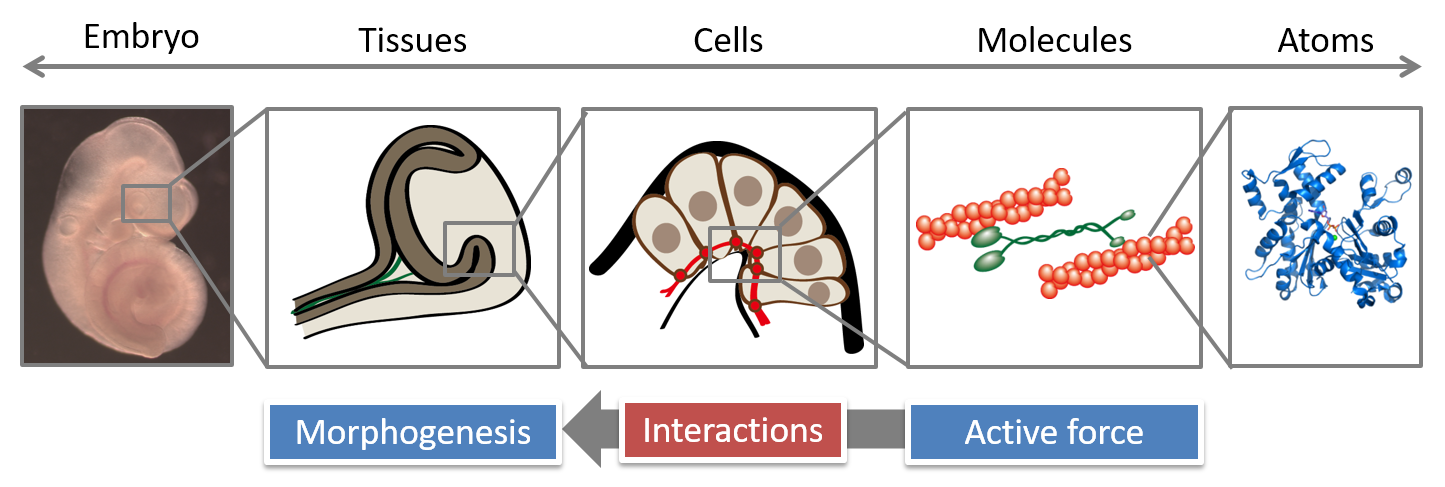
3Dバーテックスモデルは,このような多細胞の三次元的な運動・変形を,力学法則に基づいて記述する.これにより,仮定した1細胞の振る舞いから,物理法則の下で生じ得る多細胞動態を予測できる.
*開発の過程については
「バーテックスモデルの開発の歴史」を参照
多細胞組織のパッキング構造とその記述
数理モデルでは,構造や動態を記述するため,物理的な仮定を導入することにより,その構造や動態を粗視化(coarse grain)する.バーテックスモデルでは,
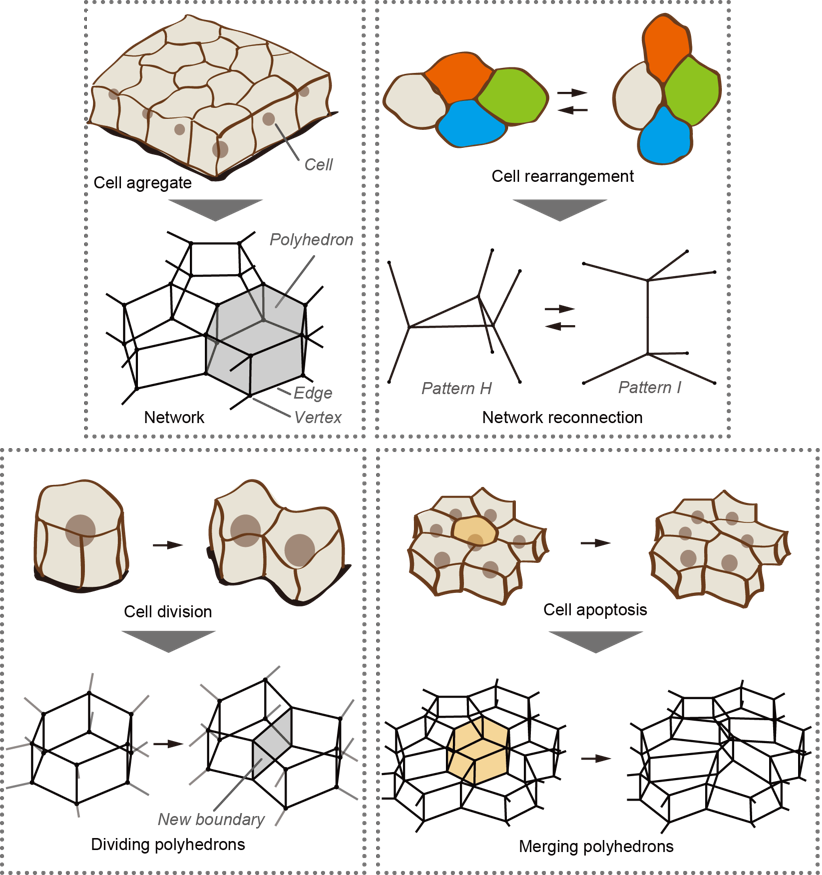
仮定1: 組織の構造は細胞が密に詰まったパッキング構造(packing geometry)であると見なし,隣接する2細胞間の境界面(boundary face)を組織の素構造とする.
これにより,個々の細胞の形態は多面体で表現される.また,境界面とその頂点(vertex)と稜線(edge)は,隣接する2細胞により共有され,組織全体は1つのネットワークにより表現される.このように,バーテックスモデルは,上皮組織や細胞塊などのパッキング構造の有する組織を対象とする数理モデルである.
1細胞レベルの3自由度とその記述
多細胞組織の変形は多様である.例えば,シート状の上皮組織の変形過程の場合,組織の伸長・曲げ,ねじれ,管腔化,分岐,多層化などが見られる.このような組織の変形は,細胞の変形・移動・分裂・細胞死など,1細胞レベルの自由度に分解できる.
- 細胞レベルの運動の自由度 → 「細胞の形の変化」+「細胞の配置の変化」+「細胞の数の変化」
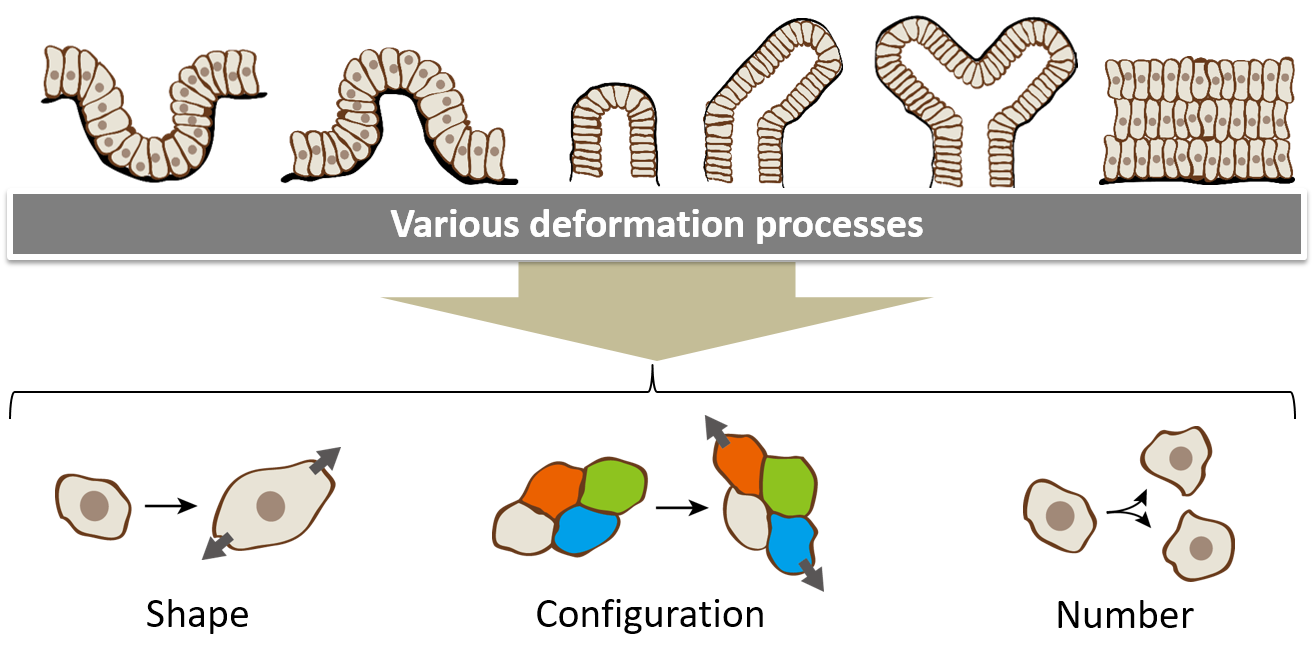
バーテックスモデルでは,この1細胞レベルの3自由度を,力学エネルギーの変化を伴う「力学的な変化」,および,力学エネルギーの変化を伴わない「幾何学的な変化」の2種類に分けて,下記のように表現する.
- 「細胞の形の変化」→「力学的な変化」
-
「細胞の形の変化」は,運動方程式に従って,頂点の運動により記述される.
- 「細胞の配置の変化」→「幾何学的な変化」
-
「細胞の配置の変化」は,細胞の隣接関係を変更する局所的なネットワーク変換により表現される.
- 「細胞の数の変化」→「幾何学的な変化」
-
「細胞の数の変化」は,細胞分裂と細胞死に相当する.細胞分裂は,1細胞に相当する多面体を分割するネットワーク変換により記述される.同様に,細胞死は,1細胞に相当する多面体を融合するネットワーク変換により記述される.
分子レベルの動態の記述
分子レベルの動態の記述は,大きく分けて,力学エネルギーに直接寄与する動態,および,寄与しないの動態の2種類に分けて導入される.
- 力学エネルギーに直接寄与する分子動態 → 「細胞の形の変化」を記述する運動方程式を通して導入
-
例えば,アクトミオシンによる力発生,細胞質の圧縮性,細胞間の接着性,細胞膜の粘弾性など.
- 力学エネルギーに直接寄与しない分子動態 → 密度などの場の量の変化として導入
-
例えば,カルシウムやモルフォゲン等の拡散性因子や遺伝子・RNAの発現量など.
「力学的な変化」としての「細胞の形の変化」の記述
物体運動を数理モデル化するためには,その運動の時間・空間スケールを考える必要がある.細胞・組織の構造物の空間スケールは原子のものに比べて非常に大きく,細胞運動の時間スケールは非常に遅い.したがって,細胞運動はニュートン力学における運動方程式に従う.マクロな物体の運動方程式は,慣性力・粘性力・外力の釣合方程式である.近年のバーテックスモデルでは,この慣性力・粘性力に対して,以下の仮定を導入し,その運動を粗視化する.
仮定2-1: 細胞運動は慣性力の影響が無視できる過程であると見なす.
もしくは,
仮定2-2: 細胞運動は慣性力と粘性力の影響が無視できる過程(準静的過程)であると見なす.
仮定2-1の場合,頂点の運動は,粘性力と外力が釣り合う過減衰の運動方程式により解かれる.仮定2-2の場合,頂点の運動は,外力のみの釣合方程式を満たす局所平衡状態の遷移過程として解かれる.また,仮定2-1では細胞内外の粘性効果が導入されるが,一方で,仮定2-2では細胞内外の粘性効果は無視される.
「幾何学的な変化」としての「細胞の配置の変化」と「細胞の数の変化」の記述
バーテックスモデルは,多細胞の幾何構造変化をネットワーク変換により記述している.このネットワーク変換を記述するため,いくつかの変換ルールが導入されている.したがって,バーテックスモデルが多細胞動態を正しく記述するためには,ネットワーク変換が物理法則に基づく仮定2-1,もしくは,仮定2-2を満たす必要がある.
個々の細胞は,組織内において自由に運動できる可能性を有する,すなわち,組織内の細胞の配置は,任意の変換が可能である.したがって,バーテックスモデルにおける幾何学的なネットワーク変換は,下記の要請を満たす必要がある.
要請1:ネットワークの変換により,仮定1に基づいて定義される組織の構造において,任意の細胞配置の変換が可能である.
さらに,ネットワークの変換は,任意の時刻において系に行われる操作である.そのため,細胞動態が仮定2-1,もしくは,仮定2-2を満たすためには,下記の要請を満たす必要がある.
要請2:ネットワークの変換過程は,エネルギーの散逸を生じない準静的過程(可逆過程)である.
ネットワーク変換のルールは,上記の要請1と要請2を満たすように設計される.ただし,実現象における細胞分裂とアポトーシスは,エネルギー変化を伴う不可逆過程なので,数理モデル上では簡単化して表現することもある.
*要請1については,京都大学の橋本敦先生と電気通信大学の長尾篤樹先生にご協力いただき,グラフ理論に基づく数学的なアプローチを使って,これを確かめた[11].その結果,仮定1において,全ての幾何構造変化を網羅できる変換ルールを見つけた.また,要請2については,ネットワーク変換の前後において力学エネルギーが保存される変換ルールを見つけた[12].
*頂点の運動の解法は,基本的な粒子系モデルと同様なので難しくない.しかし,3Dのネットワーク変換が少し複雑なため,実装には一手間いるかもしれない(
「3Dバーテックスモデルのソフトウェア公開」も参照).




